GWAS로 배우는 유전통계학 - 2.3 게놈 데이터의 품질평가
2012-04-03
Genome-wide association analysis
Identity by decent
Minor allele frequency
품질평가
2.3 게놈 데이터의 품질평가
게놈 정보는 유전계승형식을 이용한 품질평가가 중요합니다. 이번 포스팅에서는 앞서 소개한 하디-바인베르크 평형, 집단의 구조화 평가 이외의 품질평가 방법을 알아보도록 하겠습니다.
Call Rate
하나의 SNP를 다수의 개체에 대해 유전자형을 조사했을 때 어떠한 형태로든 유전자형이 결정된 개체의 비율을 SNP 당 Call Rate(CR)이라 합니다. SNP 당 CR이 낮은 유전자좌는 유전자형의 결정이 곤란하다는 것을 의미합니다. 바꿔 말하면 그 SNP좌위의 관측결과에 대한 신뢰성이 낮다는 것을 의미하는 거죠. 일반적으로 SNP 당 CR이 0.95 이상의 SNP좌위를 분석대상으로 삼습니다.
또한, 한 명의 개체에 주목하여 유전자형이 결정된 SNP의 비율인 개체당 CR도 생각하여야 합니다. 개체 당 CR이 0.99 미만의 개체는 원칙적으로 분석대상에서 제외하게 됩니다.
Minor Allele 빈도
기본적으로 GWAS는 “Common disease common variant” 가설을 전제로 합니다. 즉, 진화의 관점에서 볼 때 유전과 관계하는 흔한 질병의 원인이 되는 변이는 가계가 서로 달라도 유전정보 전체 있어서 비교적 빈도가 높은 변이일 것이라는 가설입니다. 이 가설에 따라 집단에서 어느 정도 이상 존재하는 SNP좌위 중에서 형질과 관련이 있는 유전자 좌를 탐색하는 것이 GWAS의 목적입니다. 따라서 DNA chip으로부터 관측된 모든 SNP좌위가 분석대상이 되는 것은 아닙니다. 왜냐하면, 광범위하게 발견하는 것이 곤란한 아주 드문 변이(rare variant)에 대해서는 GWAS에 의한 탐색이 어렵기 때문입니다. 따라서 하나의 SNP좌위에 2개의 allele가 있다고 했을 때 그 빈도가 작은 minor allele 빈도(minor allele frequency; MAF)가 일정한 한계 값(질병 연구에서는 보통 0.01, PGx 연구에서는 0.001) 보다 큰 SNP좌위를 분석대상으로 삼습니다.
X 염색체의 이형접합체 빈도를 이용한 성별평가
남자는 X 염색체를 하나만 가지고 있기 때문에 X 염색체의 이형 접합체(heterozygote) 빈도는 0이 되어야 합니다. 그런데 실제로는 X 염색체의 말단(telomere)에 가까운 부근에 Y 염색체와 상동 부분인 거짓 상동염색체 영역(pseudoautosomal region)에서는 이형접합체가 될 수도 있기 때문에 반드시 0이 되지 알을 때도 있습니다. 품질평가에서는 유전자형으로부터 성별의 추정하고 실제 기록과의 일치성을 조사합니다. 경험적으로 남성 X 염색체의 이형접합체 빈도가 0.2 이상의 값을 가지는 개체는 분석 대상에서 제외합니다. 단, 여성은 이 방법으로 성별평가를 할 수 없음에 주의해야 합니다.
집단에 동일 개체가 섞여 있는가에 대한 평가
관측 데이터에 동일 개체 혹은 혈연관계가 있는 개체가 포함되어 있는가는 NIBD(number of identity by decent)값을 추정해 보는 것으로 평가할 수 있습니다. IBD는 아래 그림에서 보는 것과 같이 부모로부터 남매에게 하나씩의 allele가 전달되는데 남매를 비교했을 때, 같은 종류의 allele가 몇 개 존재하는가에 대한 지표입니다.
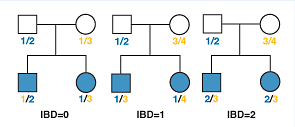
왼쪽 그림과 같이 남매 사이에 같은 종류의 allele가 없을 때는 NIBD=0, 가운데 그림은 파란색 allele 1을 남매가 공통으로 가지고 있으므로 NIBD=1, 오른쪽은 남매 모두 아버지로부터 파란색 allele 2를 어머니로부터 노란색 allele 3을 공통으로 물려받았으므로 NIBD=2가 됩니다. 일반적으로 부모와 자식 간의 BIBD=1, 일란성 쌍둥이의 NIBD=2, 혈연관계가 전혀 없을 때는 NIBD=0이 됩니다.
게놈 데이터의 품질평가에서는 모든 두 개체의 조합에 대해서 추정한 NIBD 값이 1.6 이상(최대값을 1로 했을 때 0.8 이상)의 값을 가지면 동일 개체라고 판단하고 어느 한 쪽의 개체를 분석 대상에서 제외하게 됩니다. 단, IBD를 추정할 때 적절한 SNP를 선택하기 위해 (1) SNP의 CR, (2) SNP의 MAF, (3) HWE 법칙에의 적합도 검정 유의확률(p-value)에 대해 한계 값을 설정해 조건을 만족한 SNP의 유전자형을 NIBD 추정에 이용합니다.
지금까지 3회에 걸쳐 게놈 데이터의 품질평가 방법에 대해 알아보았습니다. 실제로 이 과정이 전체 분석 시간의 절반 이상을 차지합니다. 특히, 주성분분석을 이용한 집단 구조화 평가, NIBD의 추정은 계산에 많은 시간이 걸립니다. 어떠한 분석이라도 이용하는 데이터에 오류가 있다면 분석 결과에도 오류가 포함될 가능성이 높으므로 데이터에 대한 올바른 품질평가는 분석 전체의 성패를 좌우하는 매우 중요한 과정입니다.
다음 포스팅에서는 실제 GWAS를 수행 하기위한 연구방법과 검정방법에 대해 알아보도록 하겠습니다.
참고문헌
- Balding D.J. (2006), Nature reviews Genetics, 7, 10, 781-791.
- Kruglyak L. (2008), Nature reviews Genetics, 9, 4, 314-318.
- 鎌谷直之 (2007) 遺伝統計学入門, 岩波書店 (카마타니 나오유키 (2007), 유전통계학 입문, 이와나미서점 )