한·중·일 PubMed 논문 등록 수 비교
2012-12-23
PubMed
R
Visualization
PubMed는 미국 국립 의학도서관의 국립생물공학정보센터(NCBI)가 운영하는 의학, 생물학분야의 학술논문 검색 서비스입니다. 이 데이터베이스에는 약 2,200만 건의 생물의학 논문에 대한 인용정보가 들어있고 2011년 한 해에만 986,427편의 논문에 대한 정보가 수록되어 있다고 하니 그 규모를 상상하실 수 있을 것 같습니다. 만약 자신이 의학, 생물학분야의 연구주제를 정하고 선행연구에 대한 자료를 수집하기 위해서는 반드시 들여다보아야 할 데이터베이스라 할 수 있습니다.
일본에는 이 PubMed 사용법에 관한 책도 십 수종이 있는데 이 글을 작성하면서 한국 서점 사이트를 통해 검색해 보았지만, 우리말로 된 책은 한 권도 없더군요. 사용법이 너무 쉬워서 정리된 자료에 대한 필요성을 못 느끼는지 인터넷 정보만으로도 충분한지 잘 모르겠습니다만 적어도 한두 권 정도는 우리말로 된 책이 있었으면 하는 아쉬움이 남습니다.
그럼 어느 나라가 PubMed에 가장 많은 논문을 등록했을까요?
rpsychologist.com에서는 R과 일러스트레이터를 이용해 2011년 한 해 동안 202개국 중에서 가장 많은 논문을 등록한 나라를 조사하고 이를 멋지게 시각화했습니다. 해당 블로그의 포스트를 보면 알 수 있듯이 가장 많은 논문을 등록한 나라는 역시 미국이군요. 그다음이 중국, 영국, 일본 순이네요. 우리나라는 11번째로 상위 20위 안에 들어가 있습니다.
그리고 고맙게도 이 포스트를 작성하면서 사용한 R code를 공개했습니다. 그래서 저도 이 R code를 이용해서 한·중·일 3국의 1990~2010년 PubMed 논문 10,000건당 등록건수를 조사해 보았습니다. 먼저 rpsychologist.com에서 공개한 R code는 이곳에서 구할 수 있고, 제가 그래프를 그리면서 사용한 R code는 다음과 같습니다.
> # Please read PubMed E-utility Usage Guidelines here:
> # http://www.ncbi.nlm.nih.gov/books/NBK25497/
> #
> # Check for updates and read more about this script at:
> # http://rpsychologist.com
> #
> # Packages used: 'XML', 'Rcurl' and 'plyr'
>
>
> # -----------Run these lines first--------------------- You have to set
> # working directory to were scripts are located ###
> # setwd('/path/do/directory/')
>
> # Script source
> source("PubMedTrend.R")
> # -----------------------------------------------------
>
> # Example Search Query
> query <- c(Korea = "Korea[ad]", Japan = "Japan[ad]", China = "China[ad]")
>
> # Run the script
>
> df <- PubMedTrend(query, yrStart = 1990, yrMax = 2011)
>
> # Run this to get the total hits for each query in 'query' Specify if you
> # want to get the whole 'query' or just the 'names' enter no argument if
> # you want booth 'query' and 'name'.
> df.hits <- PubTotalHits()
> df.hits <- PubTotalHits("query")
> df.hits <- PubTotalHits("names")
>
> ### EXAMPLE PLOTS ###
>
> # Note: ------ These plots wont work if you only have 1 query.
>
> library("ggplot2")
> library("directlabels")
>
> # SMOOTHED
> p <- ggplot(df, aes(year, relative, group = .id, color = .id)) +
+ stat_smooth(size = 2, span = 0.3, se = F, show_guide = F) +
+ xlab("Publication year") +
+ ylab("Publications per 10,000 PubMed articles") +
+ opts(title = paste("Pubmed hits (smoothed) for",
+ paste(names(query), collapse = ", "))) +
+ xlim(1990, 2010) + theme_bw()
>
> direct.label(p, "last.bumpup")저도 R에서 작성한 그래프를 일러스트레이터에서 약간 편집해 다음과 같은 결과를 얻었습니다.
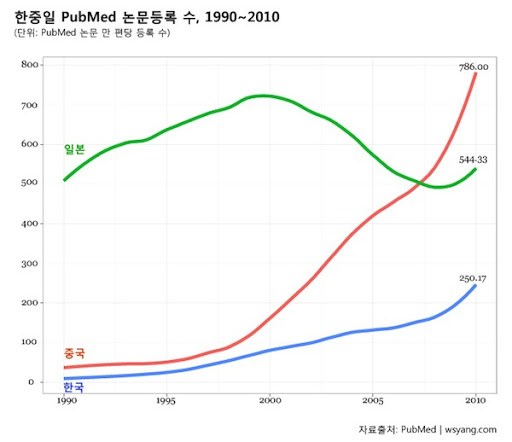
한·중·일 PubMed 논문 등록 수 비교
최근의 결과를 보니 중국은 넘사벽으로 가고 있고, 일본은 다시 정신 차리고 분발하고 있는 것 같고, 우리나라는 아직 중국, 일본 두 나라에 비해 많은 차이가 있지만, 열심히 따라가고 있습니다. 일본 선생님께 이 그래프를 보여 드리니 2000년대 초반에 일본의 논문 수가 줄어든 것은 그 시절에 국가에서 지급되는 연구비가 많이 줄었는데 그 영향이 있지 않았나 싶다고 하더군요.
이제 우리나라는 이공계 DNA를 가지신 차기 대통령 당선자가 과학기술 우대 정책을 공약으로 내 새웠으니 무언가 변화가 있겠지요. 그런데 DNA는 부모에게 물려받는 것이지 후천적으로 획득하는 것은 아니지요. 획득형질유전을 주장하는 것은 스탈린 시대의 Lysenko 주의 아닌가요?